主成分分析
|字数总计:1.2k|阅读时长:4分钟|阅读量:|
在多元统计分析中,主成分分析(Principal components analysis,PCA)是一种分析、简化数据集的技术。主成分分析经常用于减少数据集的维数,同时保持数据集中的对方差贡献最大的特征。这是通过保留低阶主成分,忽略高阶主成分做到的。这样低阶成分往往能够保留住数据的最重要方面。但是,这也不是一定的,要视具体应用而定。由于主成分分析依赖所给数据,所以数据的准确性对分析结果影响很大。
主成分分析由卡尔·皮尔逊于1901年发明,用于分析数据及建立数理模型。其方法主要是通过对协方差矩阵进行特征分解,以得出数据的主成分(即特征向量)与它们的权值(即特征值)。PCA是最简单的以特征量分析多元统计分布的方法。其结果可以理解为对原数据中的方差做出解释:哪一个方向上的数据值对方差的影响最大?换而言之,PCA提供了一种降低数据维度的有效办法;如果分析者在原数据中除掉最小的特征值所对应的成分,那么所得的低维度数据必定是最优化的(也即,这样降低维度必定是失去讯息最少的方法)。主成分分析在分析复杂数据时尤为有用,比如人脸识别。
PCA是最简单的以特征量分析多元统计分布的方法。通常情况下,这种运算可以被看作是揭露数据的内部结构,从而更好的解释数据的变量的方法。如果一个多元数据集能够在一个高维数据空间坐标系中被显现出来,那么PCA就能够提供一幅比较低维度的图像,这幅图像即为在讯息最多的点上原对象的一个‘投影’。这样就可以利用少量的主成分使得数据的维度降低了。
PCA跟因子分析密切相关,并且已经有很多混合这两种分析的统计包。而真实要素分析则是假定底层结构,求得微小差异矩阵的特征向量。
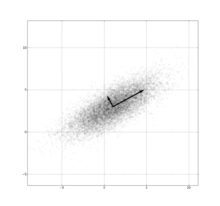
主成分分析实例:一个平均值为(1, 3)、标准差在(0.878, 0.478)方向上为3、在其正交方向为1的高斯分布。这里以黑色显示的两个向量是这个分布的协方差矩阵的特征向量,其长度按对应的特征值之平方根为比例,并且移动到以原分布的平均值为原点。应用示例
from __future__ import division
import numpy as np
import pandas as pd
class PCAcomponent(object):
def __init__(self, X, N=3):
self.X = X
self.N = N
self.variance_ratio = []
self.low_dataMat = []
def _fit(self):
X_mean = np.mean(self.X, axis=0)
dataMat = self.X - X_mean
covMat = np.cov(dataMat, rowvar=False)
eigVal, eigVect = np.linalg.eig(np.mat(covMat))
eigValInd = np.argsort(eigVal)
eigValInd = eigValInd[-1:-(self.N + 1):-1]
small_eigVect = eigVect[:, eigValInd]
self.low_dataMat = dataMat * small_eigVect
[self.variance_ratio.append(eigVal[i] / sum(eigVal)) for i in eigValInd]
return self.low_dataMat
def fit(self):
self._fit()
return self
class PCApercent(object):
def __init__(self, X, percentage=0.95):
self.X = X
self.percentage = percentage
self.variance_ratio = []
self.low_dataMat = []
def percent2n(self, eigVal):
sortVal = np.sort(eigVal)[-1::-1]
percentSum, componentNum = 0, 0
for i in sortVal:
percentSum += i
componentNum += 1
if percentSum >= sum(sortVal) * self.percentage:
break
return componentNum
def _fit(self):
X_mean = np.mean(self.X, axis=0)
dataMat = self.X - X_mean
covMat = np.cov(dataMat, rowvar=False)
eigVal, eigVect = np.linalg.eig(np.mat(covMat))
n = self.percent2n(eigVal)
eigValInd = np.argsort(eigVal)
eigValInd = eigValInd[-1:-(n + 1):-1]
n_eigVect = eigVect[:, eigValInd]
self.low_dataMat = dataMat * n_eigVect
[self.variance_ratio.append(eigVal[i] / sum(eigVal)) for i in eigValInd]
return self.low_dataMat
def fit(self):
self._fit()
return self
df = pd.read_csv(r'iris.txt', header=None)
data, label = df[range(len(df.columns) - 1)], df[[len(df.columns) - 1]]
data = np.mat(data)
print("Original dataset = {}*{}".format(data.shape[0], data.shape[1]))
pca = PCAcomponent(data, 3)
pca.fit()
print(pca.low_dataMat)
print(pca.variance_ratio)
|
原文:https://github.com/KeKe-Li/tutorial














